Was ist R?

Big Data – die zunehmend allgegenwärtige Verfügbarkeit großer und ständig wachsender Mengen an Daten ist als technisches, wirtschaftliches und gesellschaftliches Phänomen seit Jahren in der Diskussion. Das Vorhaben, aus Big Data werthaltige Informationen zu gewinnen, das sich Disziplinen wie "Data Science" oder "Analytics" auf die Fahnen geschrieben haben, erfordert leistungsfähige Statistik-Werkzeuge. Gleiches gilt für die methodisch anspruchsvolle Auswertung vergleichsweise kleiner Datenmengen, etwa im "klassischen" akademischen Bereich.
Ein Statistik-Werkzeug, das in den vergangenen Jahren immer bedeutsamer geworden ist, ist die Programmiersprache R. Anders als etwa Python, das im Bereich Data Science ebenfalls einen hohen Verbreitungsgrad genießt, ist R eine speziell für statistische Anwendungen entwickelte Sprache. Ihre Kernfunktionen liegen in der statistischen Auswertung und der Visualisierung von Daten.
R, das 1992 von Ross Ihaka und Robert Gentleman in Auckland entwickelt wurde (und deren Vornamen es wohl seinen eigenen Namen verdankt), wird als Open Source-Software unter der GNU General Public License durch die in Wien ansässige R Foundation for Statistical Computing verbreitet. Während das Herz von R durch das R (Development) Core Team (aus dem auch die Stiftung hervorgegangen ist) weiterentwickelt wird, liegt die eigentliche Stärke von R in der Verfügbarkeit von Zusatzfunktionen, in Form sogenannter Packages. Unabhängige Entwickler überall auf der Welt bieten Packages für die unterschiedlichsten Zwecke an, von der klassischen Regression bis zum Machine Learning. Mehr als 12.000 dieser Funktionspakete, die geschätzt mehr als 220.000 Funktionen beinhalten [1], stehen über das Comprehensive R Archive Network (CRAN) und einige weitere Hubs zur kostenfreien Verfügung (darunter speziell für den Bereich Bioinformatik die umfangreiche Package-Sammlung des Bioconductor-Projekts [2]). Für kaum ein statistisches Problem, und mag es auch die Anwendung einer noch so seltenen Nischen-Methode nötig machen, gibt es nicht bereits eine passende Lösung in R, die gebrauchsfertig heruntergeladen, dank der Open Source-Lizenz aber auch jederzeit an die eigenen Bedürfnisse angepasst werden kann.
So ist es eigentlich kein Wunder, dass R in der Popularität unter Data Science-Professionals mittlerweile seine kommerziellen Konkurrenten wie SAS, SPSS, Stata und Mathematica deutlich hinter sich gelassen hat [3]. Auch auf dem Arbeitsmarkt sind Bewerber mit R-Kenntnissen sehr gefragt, wie zum Beispiel die Analysen von LinkedIn-Profilen und Stellenanzeigen für Data Scientists in großen Online-Jobbörsen [4] zeigen.
Gute Gründe also, sich etwas näher mit R zu beschäftigen. Dieser Artikel bietet einen kurzen, anwendungsorientierten und einführenden Überblick über die Programmiersprache R in ihren beiden Hauptdomänen, der Analyse und der Visualisierung von Daten.
R und sein Ökosystem
Anders als manche seiner kommerziellen Konkurrenten (wie etwa SPSS) kommt R ohne schicke grafische Benutzeroberfläche daher, die das Aufrufen der statistischen Funktionen auch ohne jegliche Programmierkenntnisse erlaubt. Nach dem Download und der Installation von R (Bezug über einen der Mirror-Server des CRAN [5]), das für Windows, Linux und MacOS verfügbar ist, bietet die wenig ansehnliche RGui lediglich einige sehr rudimentäre Funktionen zum Bearbeiten und Ausführen von R-Code sowie zur Installation von Packages. Bequeme und nützliche Features wie etwa Syntax Highlighting sucht man vergeblich.
Angesichts der eher kargen Entwicklungsumgebung, mit der R von Haus aus ausgestattet ist, haben etliche andere Anbieter die Lücke geschlossen und komfortablere R-Editoren entwickelt. Als Beispiele seien hier der RCommander und RStudio genannt [6].
Der RCommander ist vor allem deshalb interessant, weil er selbst ein R-Package ist und deshalb unter dem Namen RCmdr auch von CRAN bezogen und aus der RGui heraus gestartet werden kann. Außerdem erlaubt er es, viele statistische Operationen menügesteuert aufzurufen. Die Aktionsaufrufe werden in R-Code übersetzt und ausgeführt. Dieser Ansatz kann gerade für Einsteiger interessant sein, die praxisnah verstehen wollen, wie bestimmte Operationen in R umgesetzt werden.
RStudio ist eine mächtige IDE, die von der gleichnamigen Firma grundsätzlich kostenlos (und für kommerzielle Verwendung, mit Kollaborationswerkzeugen, Sever-Integration und besserem Support kostenpflichtig) zur Verfügung gestellt wird. Das Flaggschiff-Produkt von RStudio ist nicht nur eine sehr populäre Entwicklungsumgebung für R, sondern auch ein gutes Beispiel dafür, wie sich funktionierende Geschäftsmodelle im Umfeld einer Open Source-Software realisieren lassen. Unternehmen wie RStudio, die ein Interesse an der Förderung der Entwicklung, Verbreitung und Verwendung von R (insbesondere auch im geschäftlichen Umfeld) haben, sind im R Consortium zusammengeschlossen, um Projekte, die diesen Zielen dienen, gemeinsam zu finanzieren und voranzutreiben. Zu den Mitgliedern des Konsortiums zählen unter anderem auch Microsoft, IBM und Oracle.
Möchte man keine spezielle R-Entwicklungsumgebung wie RStudio benutzen, kann man R-Code natürlich zum Beispiel auch in Notepad++, Sublime oder Microsofts kostenfreiem Visual Studio Code mit allem Komfort bearbeiten.
Wer bei der Entwicklung mit R Hilfe und fachliche Informationen sucht, wird oft fündig in den obligatorischen Dokumentationen der R-Packages, die häufig sehr ausführlich sind und stets auch ausführbare Code-Beispiele beinhalten. Die sogenannten CRAN Task Views bieten zu einem bestimmten Gebiet (beispielsweise Finance, Machine Learning oder Time Series) einen guten Überblick über die jeweils relevanten R-Packages und ihre Spezifika [7]. Über neue Entwicklungen rund um R (unter anderem auch die Veröffentlichung bedeutender Packages) berichtet die Open Access-Zeitschrift R Journal und das (nicht allein auf R beschränkte) Journal of Statistical Software [8]. Gute Anlaufstationen sind darüber hinaus natürlich auch die populären Internet-Communities wie zum Beispiel der Blog-Aggregator R-bloggers oder auch StackOverflow, wo der Anteil an R-bezogenen Threads in den vergangenen Jahren deutlich gestiegen ist [9]. Hier wird R, obwohl ja eine Special-Purpose-Language, dieses Jahr wohl unter den am häufigsten diskutierten Programmiersprachen überhaupt sein [4].
Die Sprache R – Alles Objekt, oder was?
In diesem Abschnitt werden die wichtigsten R-Sprachkonzepte vorgestellt. Der Fokus liegt dabei auf Themen, die für die praktische Arbeit mit R besonders wichtig sind. Zugunsten dieser wird auf eine breite Diskussion fortgeschrittener Themen, wie beispielsweise Environments (dem ausgefuchsten Namespace-Konzept von R) an dieser Stelle verzichtet.
Ausführungsmodi
R ist standardmäßig eine interpretierte Sprache, es existiert aber auch ein Bytecode-Compiler [10]. Anders als in den meisten anderen Programmiersprachen (aber ähnlich zu anderen Statistiksprachen wie etwa Stata) kann es in R Sinn machen, Anweisungen nicht nur in Form von Programmen auszuführen, also im Skriptmodus, sondern auch im interaktiven Modus, das heißt, durch Eingabe einzelner Anweisungen in das Prompt, das in R als > angezeigt wird. Auf diese Weise können zum Beispiel live die Daten "befragt" werden oder an den optimalen Einstellungen für eine Grafik experimentiert werden. Die zuletzt ausgeführten Anweisungen können jederzeit mit Hilfe der Funktion history() eingesehen werden.
Während R-Codedateien reine Textdateien sind, bietet R die Möglichkeit, R-Objekte (zum Beispiel Datensätze oder Details statistischer Schätzmodelle) mit Hilfe der Funktion save() im binären .RData-Format zu speichern. Mit save.image() kann der gesamte R-Workspace gesichert werden. Auf diese Weise kann man die Arbeit zu einem späteren Zeitpunkt an exakt der Stelle wiederaufnehmen, wo man sie verlassen hat.
Eine verhältnismäßig neue Art, R-Code auszuführen, ist in Form serverseitiger Apps. Dazu wird das Package shiny verwendet, das von RStudio kostenfrei zur Verfügung gestellt wird [11]. Es erlaubt, webbasierte Anwendungen aus vollständig über R-Code gesteuerten Ein- und Ausgabe-Elementen zu erzeugen. Diese Apps können dann entweder auf einem eigenen, shiny-fähigen Linux-Server (ein entsprechender Server ist in einer Basisversion als Open Source-Lösung erhältlich) oder über die RStudio-Plattform shinyapps.io (kostenlos für bis zu 5 Apps mit zusammen höchstens 25 Stunden Rechenzeit pro Monat) gehostet werden. Auf diese Weise können R-Programme, die über Standardeingabeelemente wie Schieberegler und Textboxen parametrierbar sind, über das Web Benutzern zugänglich gemacht werden, ohne dass diese selbst R installiert haben oder R-Code verstehen müßten.
Objektbasierung versus Objektorientierung
In R dreht sich alles um Objekte. Wie John Chambers, Entwickler der R-Vorgänger-Sprache S es in Bezug auf R auf den Punkt gebracht hat: "Everything that exists is an object. Everything that happens is a function call".
Nicht nur Variablen, auch Funktionen, Operatoren und ganze Ausdrücke, aus denen ein R-Skript aufgebaut ist, sind Objekte. Dennoch ist R keine vollständig objektorientierte Sprache im klassischen Sinne, wie etwa Java. Man kann ganz wunderbar mit R arbeiten, ohne zu verstehen, was Konzepte wie Klassen, Vererbung, Kapselung und Polymorphismus bedeuten.
R unterstützt unterschiedlich "scharfe" Ansätze der Objektorientierung, die zu betrachten über die Zielsetzung dieses Artikels hinausginge. Den Feinschmeckern unter den Lesern sei bzgl. der Anwendung des objektorientierten und des funktionalen Programmierparadigmas in R das aufschlussreiche Papier von John Chambers empfohlen, dem auch das einprägsame Zitat entnommen ist [12].
Grundlegendes zur Syntax
R-Programme bestehen aus Ausdrücken, zum Beispiel Zuweisungen oder Funktionsaufrufen. Ein Ausdruck schließt mit dem Zeilenende ab. Nur wenn mehr als ein Ausdruck in einer Zeile steht, müssen die Ausdrücke durch Semikola voneinander getrennt werden.
Kommentare werden durch # eingeleitet und reichen stets bis zum Zeilenende; mehrzeilige Kommentare kennt R nicht.
R ist case-sensitive. Für Objektnamen (zum Beispiel von Funktionen und Variablen) sind neben den alphanumerischen Zeichen auch der Punkt und der Unterstrich erlaubt. Objektnamen mit Unterstrich sind allerdings eher selten anzutreffen, häufiger wird der Punkt benutzt, um Objektbezeichner zu strukturieren.
Codeblöcke (etwa in Schleifen oder Funktionen) werden wie in C mit geschweiften Klammern umschlossen. Überhaupt finden sich Entwickler mit C-Erfahrung gut in R zurecht, da die Syntax (etwa bei Bedingungen) teilweise analog zu C aufgebaut ist. Auch kommen die aus C bekannten logischen und Vergleichsoperatoren wie &&, || oder != zum Einsatz.
Orientierende Richtlinien für die verständliche Gestaltung von Code bietet beispielsweise Google’s R Code Style Guide [13].
Variablen in R
Variablen in R müssen nicht deklariert werden, sondern werden im Moment einer Zuweisung dynamisch erschaffen. Zugegriffen wird auf Variablen über deren Namen, der in R als Symbol bezeichnet wird. Eine Wertezuweisung erfolgt in R mit Hilfe des Zuweisungsoperators <-; im folgenden wird beispielhaft einer Variablen x der Wert 5 zugewiesen, genauer gesagt, es wird ein Objekt mit dem Wert 5 geschaffen, auf das über das Symbol x zugegriffen werden kann:
x <- 5
Als Zuweisungsoperator kann auch das Gleichheitszeichen verwendet werden. Der Pfeiloperator hat allerdings den Vorteil, dass er die Richtung der Zuweisung anzeigt. Objekt und zugewiesener Wert können deshalb auch vertauscht werden: 5 -> x ist demnach eine gültige Zuweisung. Der Zuweisungsoperator ist übrigens eigentlich eine Funktion (und diese wiederum ein Objekt, weil einfach alles ein Objekt ist). Wir erinnern uns an den eingangs dieses Abschnitts zitierten John Chambers: "Everything that happens is a function call". Der Funktionsaufruf `<-`(x,5), hat die gleiche Wirkung wie die obige Zuweisung und ist die Operation, die der R-Interpreter beim Aufruf unserer Zuweisung durchführt.
Die wichtigsten elementaren Datentypen in R sind:
- integer (Ganzzahlen),
- double (Gleitkommazahlen),
- logical (logische Werte, TRUE und FALSE) und
- character (Zeichenketten).
Werte gleichen Typs lassen sich mit Hilfe der Funktion c() zu sogenannten Vektoren zusammensetzen:
> v <- c(1,2,3,5) > v [1] 1 2 3 5
Bei Eingabe des Variablennamens am Prompt (>) zeigt R den Inhalt der Variable an (präziser: den Inhalt des Objekts, das mit dem eingegebenen Symbol angesprochen wird). Auf die Elemente eines Vektors kann durch Indizierung in eckigen Klammern zugegriffen werden, wobei die Indizierung bei 1 startet:
> v[4] [1] 5
Die Zahl in eckigen Klammern vor dem R-Output ist der Index des ersten Elements, das in der Zeile angezeigt wird (eine nützliche Information, wenn etwa lange Vektoren ausgegeben werden).
Komplexere Datenkonstrukte, die auch Daten unterschiedlicher Typen aufnehmen können, sind Listen (Typ list) und Dataframes (Typ data.frame). Im folgenden Beispiel werden zwei Objekte erzeugt, ein Vektor aus Zeichenketten und ein Vektor mit einer Zahl als einzigem Element, und dann zu einer Liste zusammengefügt:
> x1 <- c("Waldi", "Hasso")
> x2 <- c(42.5)
> meine.liste <- list(Hund=x1, SchulterMittel=x2)
> meine.liste
$Hund
[1] "Waldi" "Hasso"
$SchulterMittel
[1] 14.5
> meine.liste$Hund[2]
[1] "Hasso"Beim Aufruf der Funktion list() können optional die Namen der Elemente angegeben werden. Über diese Namen können die Elemente dann in der Notation liste$elementname angesprochen werden. Alternativ kann ein Element über seinen Index als liste[[index]] adressiert werden.
Während Listen oft von statistischen Funktionen verwendet werden, um mehrere Werte zurückzugeben, sind Dataframes das Workhorse der Statistik in R, denn sie bilden Tabellen ab. Eine Möglichkeit, Dataframes zu erzeugen, besteht darin, mehrere (anders als bei Listen notwendigerweise gleich lange) Vektoren miteinander zu verbinden:
name <- c("Katharina", "Peter", "Sophie", "Anna", "Joachim")
geschlecht <- c("w", "m", "w", "w", "m")
alter <- c(18, 25, 28, 22, 37)
freunde <- data.frame(name, geschlecht, alter)Um sich den Inhalt des Dataframes anzuschauen, genügt es wiederum, seinen Namen am Prompt einzugeben. Alternativ können die Daten mit der Funktion View() in einer Spreadsheet-artigen Darstellung in der R-Entwicklungsumgebung angezeigt werden.
Auf die Spalten eines Dataframes kann nun in der Notation dataframe$spalte, also zum Beispiel freunde$geschlecht zugegriffen werden. Soll ein Datenelement eines Dataframes über Indizes adressiert werden, ist eine Notation der Form dataframe[zeile,spalte] möglich, also beispielsweise freunde[1,2], was den Wert der zweiten Variable (=Spalte) im ersten Datensatz (=Zeile) liefert, in diesem Beispiel also w, das Geschlecht von Katharina. Indizes können auch weggelassen werden und so zum Beispiel mit freunde[,3] die dritte Spalte des Dataframes, also die Variable alter, als Vektor angesprochen werden.
Neben den elementaren Datentypen sowie den Vektoren, Listen und Dataframes gibt es noch eine ganze Reihe weiterer Objektarten in R, beispielsweise factors. Das sind kategoriale Variablen mit einer vordefinierten Menge an Ausprägungen, beispielsweise Schulnoten oder Haarfarben. Oder für Funktionen (die ja ebenfalls Objekte sind) die Datentypen special und closure, je nachdem ob die Funktion fest in R eingebaut ist (wie beispielsweise die Funktion `<-`, die den Zuweisungsoperator implementiert) oder nicht. Mit benutzerdefinierten Funktionen beschäftigen wir uns unten noch etwas ausführlicher.
R ist schwach typisiert: Wie an den obigen Beispielen gesehen, muss der Objekttyp beim Initialisieren von Variablen nicht angegeben werden (eine Deklaration, bei der der Typ angegeben werden könnte, gibt es ja ohnehin nicht). Stattdessen bestimmt R den Typ selbst. Dabei kommt ein als Coercion bezeichneter Mechanismus zur Anwendung. Coercion versucht sicherzustellen, dass Daten dem für die jeweils durchgeführte Operation notwendigen Typ entsprechen. Werden beispielsweise Zahlen und Text-Informationen gemeinsam in einem Vektor zusammengeführt, erhält dieser Vektor automatisch den Typ character, damit beide Typen von Daten abgelegt werden können. R wählt also vereinfacht gesagt den kleinsten gemeinsamen (Typen-)Nenner.
Alle Daten, die R benötigt, werden im Speicher gehalten. Um den Speicherbedarf zu managen, benutzt R automatische Entsorgung nicht mehr benötigter Daten (Garbage Collection) und bedarfsgesteuertes Datenladen (Lazy Loading).
Kontrollstrukturen
Innovationen im Bereich der Kontrollstrukturen sind in Programmiersprachen bekanntlich selten, und so kennt auch R das klassische Steuerkonstrukt if/else, dessen Syntax analog zu C ist:
if(Bedingung) { Anweisungsblock } else { Anweisungsblock }Die for-Schleife in R unterscheidet sich syntaktisch von ihrem Namensvetter in C. In R hat sie die Form
for (variable in liste) { Anweisungsblock }Eine "typische" for-Schleife würde durch einen Vektor mit ganzzahligen Elementen iterieren; da liste aber eine beliebige Liste (und damit auch eine Liste von Objekten ganz unterschiedlichen Typs) sein kann, ist es in R sehr einfach, mit for die gleiche Wirkung zu erzielen, für die man in anderen Sprachen eine for each-Schleife bemühen würde.
Neben der klassischen while-Schleife der Form
while (Bedingung) { Anweisungsblock }existiert eine repeat-Schleife ohne weitere Laufbedingungen, die einfach so lange läuft, bis sie mit break verlassen wird.
Bei allen Kontrollstrukturen können die geschweiften Klammern entfallen, wenn nur ein einziger Ausdruck folgt.
Funktionen
Wie alles in R sind auch Funktionen Objekte. Sie werden durch eine Zuweisung von Funktionskopf und -rumpf zu einem Symbol erzeugt, zum Beispiel:
quadrat <- function(x)
{
return(x^2)
}Der schwachen Typisierung in R entsprechend werden die Argumente im Funktionskopf ohne Datentyp angegeben.
Funktionen müssen stets mit runden Klammern aufgerufen werden, auch wenn keine Argumente übergeben werden; anderenfalls wird der Quelltext der Funktion angezeigt (ein durchaus praktisches Feature).
Die Argumente einer Funktion können mit einem Standardwert versehen werden, zum Beispiel quadrat<-function(x=3), und über ihren Namen angesprochen werden: quadrat(x=4) ist also ein gültiger Funktionsaufruf. Funktionen können auch eine unbestimmte Zahl von Argumenten erwarten, wie im folgenden Beispiel, in dem die Summe eine Reihe von zuvor quadrierten Argumenten berechnet wird:
quadratsumme <- function(...)
{
x <- as.double(list(...))
return(sum(x^2))
}Hier wird aus dem speziellen Objekt …, das eine undefinierte Zahl von Argumenten darstellt, zunächst eine Liste gemacht und diese dann in einen Vektor von Fließkommazahlen umgewandelt, mit dem dann weitergearbeitet werden kann. Nun könnte die Funktion etwa als quadratsumme(3,4.05) aufgerufen werden.
Wenn einer R-Funktion ein Argument übergeben wird, erzeugt der Interpreter zunächst ein neues Environment (Namensraum) für die Funktion und dann ein Objekt, das mit dem Wert des Arguments initialisiert wird; Argumentübergaben erfolgen in R also de facto "by value". Da Funktionen selbst Objekte sind, können sie anderen Funktionen auch als Argument übergeben werden. Ein Beispiel dafür ist die Funktion tapply, die wir uns weiter unten nochmal anschauen werden.
Bestimmte Funktionen in R, wie beispielsweise print oder summary, können scheinbar Argumente von sehr unterschiedlichen Typen verarbeiten, u. a. die komplexen Rückgabeobjekte von statistischen Modellschätzungen. Solche Funktionen werden als generische Funktionen bezeichnet. Sie sind letztlich nur Hüllen-Funktionen, die in Abhängigkeit der Klasse des ihr übergebenen Arguments die entsprechende "Spezialfunktion" für die zugehörige Klasse aufrufen. Die Spezialfunktion, die die Ergebnisse einer linearen Regression darstellt, heißt beispielsweise print.lm (lm für linear model). Sie wird von der generischen Funktion print immer dann aufgerufen, wenn print ein lm-Objekt als Argument übergeben bekommt. Hier scheint einer der Ansätze durch, wie R mit Objektorientierung umgeht; nämlich, dass die Methoden nicht zum Objekt gehören, sondern über generische (Hüllen-)Funktionen realisiert werden.
Arbeiten mit Packages
Packages, die Zusatzpakete von R, die vor allem über das CRAN verfügbar sind, können mit library(packagename) geladen werden. Das Package muss dazu zunächst mit install.package(packagename,dependencies=TRUE) installiert werden. Das empfehlenswerte Argument dependencies=TRUE stellt sicher, dass gleich alle Packages, von denen das zu installierende abhängt (auf deren Funktionen es also zurückgreift) mit installiert werden, sofern nicht bereits geschehen. Eine Liste der installierten Packages kann mit installed.packages() aufgerufen werden.
Ist das Package geladen, können die darin zur Verfügung gestellten Funktionen ohne weiteres Zutun aufgerufen werden. Mit ?funktionsname kann am Prompt zudem die als Bestandteil des Packages installierte Hilfe zu einer Funktion aufgerufen werden.
Statistik mit R: Erste Schritte
Möchte man R für statistische Anwendungen nutzen, muss man offensichtlich zunächst die Daten in R verfügbar machen. R bietet standardmäßig eine ganze Reihe von Funktionen, um Daten aus textbasierten Dateien einzulesen, insbesondere die allgemeine Funktion read.table(), bei der alle wichtigen Parameter wie Spaltenseparatoren, Begrenzer für Zeichenketten und das Dezimaltrennzeichen spezifiziert werden können. Die Funktionen read.csv() (Komma als Spaltenseparator, Punkt als Dezimaltrennzeichen), read.csv2() (Semikolon als Spaltenseparator, Komma als Dezimaltrennzeichen), read.delim() (Tabulator als Spaltenseparator, Punkt als Dezimaltrennzeichen) und read.delim2() (Tabulator als Spaltenseparator, Komma als Dezimaltrennzeichen) sind letztlich spezielle Versionen von read.table() mit jeweils unterschiedlichen Standardwerten für die zentralen Format-Argumente.
Auch für Daten aus anderen Statistik-Paketen stehen etliche Import-Funktionen zur Verfügung, z. B. read.spss() (für SPSS-Daten), read.ssd() (für SAS-Daten), read.dta (für Stata-Daten); diese Funktionen sind allesamt im Package foreign enthalten. Excel-Daten (sowohl .xls als auch .xlsx) können beispielsweise mit read.xls() aus dem gdata-Package eingelesen werden.
Durch die Zuweisung
daten <- read.table("C:/umfragedaten.csv", sep=";", dec=".");könnte man nun etwa die in umfragedaten.csv enthaltene Datentabelle in einem Dataframe namens daten speichern (Achtung: R verwendet keinen Backslash!). Für das Einlesen von Daten aus dem Web muss übrigens lediglich der Dateiname durch die URL ersetzt werden.
Wir wollen es uns hier aber einfach machen und verwenden den Datensatz Anscombe, der im R-Package car enthalten ist. Viele weitere Datensätze zum Ausprobieren findet man auch im R-Package datasets.
Mit names(Anscombe) können wir uns, nachdem wir das Package mit library(car) geladen haben, die Variablen anschauen, die im Datensatz enthalten sind:
> names(Anscombe) [1] "education" "income" "young" "urban"
Die Variablen bilden für die 51 US-Bundestaaten (inkl. Washington D.C.) die Pro-Kopf-Ausgaben für Bildung (education), das Durchschnittseinkommen (income), sowie die Anzahl der Unter-Achtzehnjährigen (young) und die Anzahl der in urbanen Gebieten lebenden Bürger (urban), jeweils pro 1000 Einwohner, für das Jahr 1970 ab. Die Abkürzungen der Bundesstaaten stehen als Zeilennamen im Datensatz und können mit row.names(Anscombe) angezeigt werden. Sie werden natürlich auch angezeigt, wenn man sich den Datensatz mit View(Anscombe) vollständig darstellen läßt.
Um sich einen ersten Überblick über den Datensatz zu verschaffen, kann man sich mit head(Anscombe) und tail(Anscombe) die ersten bzw. letzten fünf Datenzeilen anschauen.
Einen systematischeren Eindruck von den Daten erhält man natürlich mit den Werkzeugen der deskriptiven Statistik; dafür bietet R unter anderem die Funktionen mean() für das arithmetische Mittel, median() für den Median (50%-Quantil), min() und max() für den kleinsten und größten Wert, und quantile() für beliebige Quantile an. Mit
> median(Anscombe$income) [1] 3257
ließe sich also zum Beispiel der Median des durchschnittlichen Einkommens aller US-Bundesstaaten ermitteln (die Zahl sieht niedrig aus, aber es gab natürlich sowohl ein erhebliches reales Wachstum als auch eine beachtliche Inflation seit 1970). Die Funktion summary(), die eine einzelne Variable (also einen Vektor) oder auch einen ganzen Datensatz als Argument übernimmt, fasst gängige Kennzahlen der deskriptiven Statistik übersichtlich zusammen.
Mit Hilfe von cor() ermittelt man den Korrelationskoeffizienten als Zusammenhangsmaß, hier am Beispiel des Zusammenhangs von Bildungsausgaben und dem Anteil junger Bürger an der Bevölkerung (die zugrundeliegenden Funktionen für Varianz und Kovarianz heißen in R var() und cov()):
> cor(Anscombe$education, Anscombe$young) [1] 0.3114855
Ein Wort noch zum Umgang mit fehlenden Daten: Viele Funktionen in R geben standardmäßig die spezielle Konstante NA (für not available) zurück, wenn die Daten, auf die sie angewendet werden, Missings, also fehlende Datenpunkte, beinhalten. So ist zum Beispiel der Wert der Summenfunktion sum() gleich NA, wenn der Vektor, dessen Elemente sie aufsummiert, ein Missing enthält. Durch Setzen des von vielen Funktionen unterstützten Arguments na.rm auf TRUE schließt man die Missings einfach aus und stellt sicher, dass die Funktionen sinnvolle Werte zurückgeben, auch wenn die Daten "löchrig" sind. Mit der Funktion is.na() kann man prüfen, wo in einem Vektor Missings vorliegen.
Die statistischen Funktionen von R lassen sich nicht nur auf ganze Vektoren oder Dataframes anwenden, sondern mit Hilfe der Funktion tapply() auch auf gruppierte Daten. So könnte man beispielsweise den Median der Bildungsausgaben über alle US-Bundesstaaten bestimmen, und zwar abhängig davon, ob der Anteil der Jugendlichen an der Bevölkerung höher oder niedriger als im Durchschnitt aller Bundesstaaten ist:
> tapply(Anscombe$education, INDEX=Anscombe$young>mean(Anscombe$young), FUN=median) FALSE TRUE 189 207
Das Argument INDEX ist dabei das Gruppierungskriterium (hier: ein Ausdruck, der einen Vektor logischer Werte zurückgibt), FUN die anzuwendende Funktion. An diesem Beispiel sieht man sehr schön, dass in R auch Funktionen, die ja selbst Objekte sind, einer anderen Funktion als Argumente übergeben werden können.
Über die deskriptive Statistik hinaus lässt sich nun das beinahe unbegrenzte Spektrum statistischer Methoden in R anwenden. An dieser Stelle nur ein einfaches Beispiel: Wollte man mit Hilfe einer linearen Regression etwas systematischer die Hypothese überprüfen, dass Bundesstaaten mit einem höheren Anteil jüngerer Bürger pro Kopf mehr für Bildung ausgeben, könnte man mit der Funktion lm() (für linear model) eine lineare Regression schätzen:
> m <- lm(education ~ income + young + urban, data=Anscombe)
> summary(m)
Call:
lm(formula = education ~ income + young + urban, data = Anscombe)
Residuals:
Min 1Q Median 3Q Max
-60.240 -15.738 -1.156 15.883 51.380
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) -2.868e+02 6.492e+01 -4.418 5.82e-05 ***
income 8.065e-02 9.299e-03 8.674 2.56e-11 ***
young 8.173e-01 1.598e-01 5.115 5.69e-06 ***
urban -1.058e-01 3.428e-02 -3.086 0.00339 **
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Residual standard error: 26.69 on 47 degrees of freedom
Multiple R-squared: 0.6896, Adjusted R-squared: 0.6698
F-statistic: 34.81 on 3 and 47 DF, p-value: 5.337e-12Wie viele statistische Funktionen gibt auch lm() ein Objekt zurück, das selbst letztlich eine Liste von Objekten ist und die Modellergebnisse abbildet.
> names(m) [1] "coefficients" "residuals" "effects" "rank" [5] "fitted.values" "assign" "qr" "df.residual" [9] "xlevels" "call" "terms" "model"
Nun könnte man etwa mit m$coefficients auf den Vektor der Regressionskoeffizienten zugreifen, um damit weiterzuarbeiten.
Grafiken mit R: Erste Schritte
R verfügt über sehr starke Grafik-Funktionalitäten, um Daten zu visualisieren. Von Haus aus wird das Standard-Grafikpaket graphics sowie das Paket lattice installiert, das insbesondere gut geeignet ist, um Grafiken mit mehreren Panels zu erzeugen.
Daneben existiert mit ggplot2 ein sehr beliebtes Grafik-Package, das einem gänzlich anderen, als "grammar of graphics" bezeichneten Ansatz folgt (daher auch das gg in ggplot2). Dabei werden Grafiken auf einer abstrakten Ebene als Mapping von Daten zu visuellen Eigenschaften der Abbildung verstanden und logisch auf dieser Grundüberlegung aufbauend beschrieben. Wer sich für die Anwendung dieses Packages, mit dem sich sehr attraktive Grafiken erzeugen lassen, interessiert, sei auf das praxisorientierte und reichlich illustrierte R Graphics Cookbook von Winston Chang verwiesen [14].
Für den Beginn aber reichen die Mittel des zur R-Standardinstallation gehörenden Packages graphics vollkommen aus. Es stellt eine ganze Reihe von Funktionen zur Verfügung, um für die Darstellung sowohl kategorialer als auch kontinuierlicher Daten unterschiedlichste Arten von Grafiken zu erzeugen, darunter Histogramme (hist()), Boxplots (boxplot()), Balkendiagramme (bar()), Kuchendiagramme (pie()), Punktewolken (plot()) oder Mosaikplots (mosaicplot()). Auch für dreidimensionale Daten existieren etliche Darstellungsfunktionen, darunter für dreidimensionale Oberflächen persp() und für Heatmaps die gleichnamige Funktion heatmap().
Jede Grafik kann durch eine gefühlt unbegrenzte Anzahl von Argumenten an jeden auch noch so ausgefallenen Wunsch des Benutzers angepasst werden. Dabei können die Parameter entweder direkt der jeweiligen Grafikfunktion übergeben werden (und gelten damit nur für die aktuelle Grafik), oder sie werden mit der Funktion par() für alle Grafiken auf dem aktuellen Grafik-Device festgelegt. Durch Umlenken des Outputs auf andere R-Grafik-Devices lassen sich Grafiken auch bequem in Formate wie BMP, JPG, PNG oder PDF exportieren.
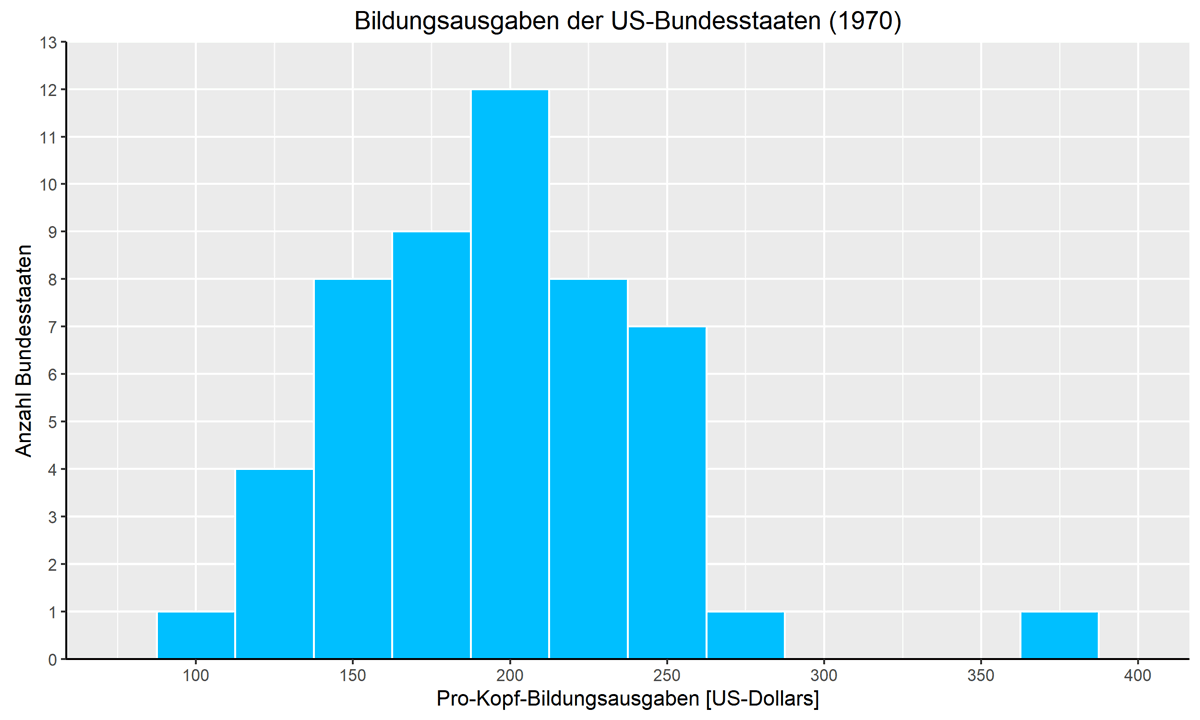
Im folgenden ein Beispiel für ein sehr einfaches Histogramm, das die Verteilung der Bildungsausgaben darstellt (der Ausreißer mit den besonders hohen Bildungsausgaben ist übrigens Alaska):
hist(Anscombe$education, breaks=seq(from=87.5, to=400, by=25),
main = "Bildungsausgaben der US-Bundesstaaten (1970)",
xlab = "Pro-Kopf-Bildungsausgaben [US-Dollars]",
ylab = "Anzahl Bundesstaaten",
col = "deepskyblue1",
border = "white")Mit dem Argument breaks wird R angewiesen, die Daten für das Histogramm in 25 Dollar breite Klassen zusammenzufassen. Alternativ hätte man die Klassengrenzen auch als Vektor expliziter Werte übergeben oder auch nur eine Anzahl von Klassen vorgeben und R deren Breite selbst bestimmen lassen können.
Mit den Argumenten col und border werden die Füll- und die Randfarbe der Balken festgelegt. R kennt standardmäßig neben hexadezimalen RGB-Farbcodes wie etwa #FF0000 für Rot auch eine Reihe von Farbkonstanten [15]. Natürlich kann R auch mit anderen Farbräumen wie HSL umgehen.
Ein ähnlich aussehendes Histogramm würde man mit dem Package ggplot2 folgendermaßen erzeugen:
ggplot(data = Anscombe, aes(x = education)) + geom_histogram(fill="deepskyblue1", colour="white", binwidth=25) + labs(title="Bildungsausgaben der US-Bundesstaaten (1970)", x="Pro-Kopf-Bildungsausgaben [US-Dollars]", y = "Anzahl Bundesstaaten") + scale_x_continuous(breaks=seq(from=0, to=400, by=50), limits=c(75,400)) + scale_y_continuous(expand = c(0, 0), breaks=seq(from=0, to=13, by=1), minor_breaks=NULL, limits=c(0,13)) + theme(axis.line.x = element_line(colour="black", size=0.5),axis.line.y = element_line(colour="black", size=0.5));
Hier sieht man sehr schön, wie entsprechend dem Konzept der grammar of graphics zunächst ein ggplot-Objekt erzeugt wird, das den Anscombe-Datensatz als Daten übergeben bekommt. Mit aes (für aesthetics) sagen wir ggplot, dass wir die x-Koordinate unserer Darstellung mit den Bildungsausgaben belegen wollen. Hier mappen wir also numerische Daten auf eine Eigenschaft der Darstellung.
Bis hierhin ist noch nicht klar, welche Art von Darstellung wir tatsächlich wählen. Das passiert erst im nächsten Schritt, in dem wir ein sogenanntes geom, d. h. eine Art der Darstellung, hinzufügen (buchstäblich "hinzuaddieren"), in unserem Beispiel ein Histogramm. Auf ähnliche Weise fügen wir schließlich Labels und eine Achse hinzu.
Fazit
Aufgrund seines Open Source-Charakters und der buchstäblich Tausenden von Zusatzpaketen ist R längst zum Schweizer Taschenmesser für Statistiker und Data Scientists geworden. Das spiegelt sich unter anderem nicht zuletzt auch in der hohen Nachfrage nach R-Kenntnissen auf dem Arbeitsmarkt und der Unmenge an R-bezogenen Tutorials, Blogs und Foren im Internet wider. Jeder, der sich für Statistik interessiert, sollte sich einmal mit R beschäftigt haben. Dieser Artikel liefert dazu eine erste Einführung in die Sprache und ihre Charakteristika.
- R. Muenchen: The Popularity of Data Science Software, 2017
- Package-Sammlung des Bioconductor-Projekts
- K. Rexer, P. Gearan and H. Allen: 2017 Data Science Survey, 2017
- D. Smith: New surveys show continued popularity of R, 2015
- Mirror-Server des CRAN
- RCommander
RStudio - CRAN Task Views
- Open-Access-Zeitschrift R Journal
J. Fox und A. Leanage: R and the Journal of Statistical Software, 2016 - StackOverflow
R-bloggers - T. Galili: Speed up your R code using a just-in-time (JIT) compiler, 2012
- Package shiny
- J. M. Chambers: Object-Oriented Programming, Functional Programming and R, Statistical Science, Vol. 29, No. 2, 167–180, 2017;
- Google’s R Code Style Guide
- W. Chang: R Graphics Cookbook, O’Reilly, 2013
- Reihe von Farbkonstanten